蛋白质组生物信息学分析服务 - 质谱定量蛋白质组数据分析
DIA数据分析流程:
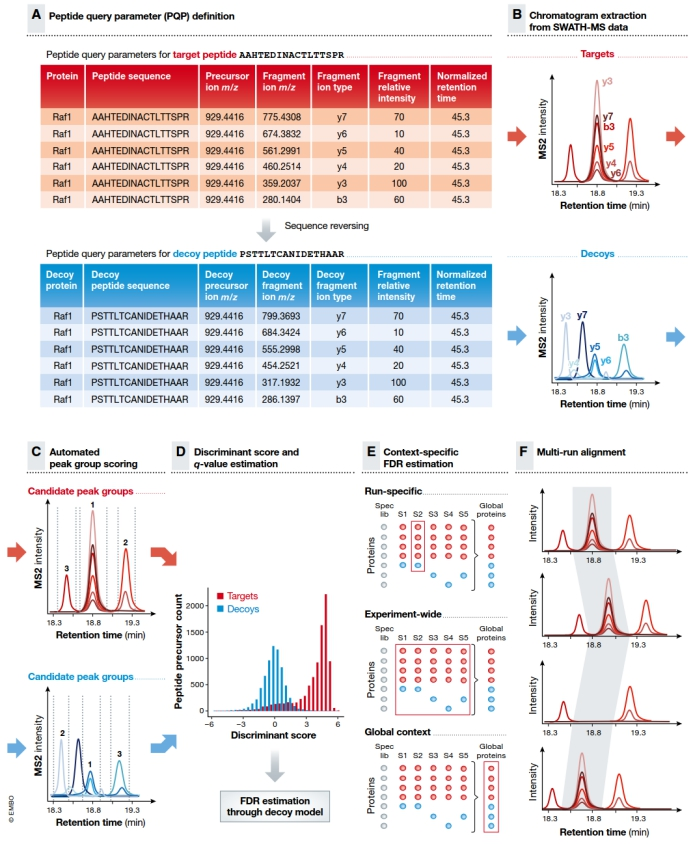
DIA的数据分析始于一组肽查询参数(peptide query parameters, PQP),这些参数包含了保留时间,前体离子质量,碎片离子质量和碎片离子靶肽的信号强度等信息。诱饵肽也需要PQP信息,诱饵肽可以通过反转氨基酸来生成PQP,同时保留末端氨基酸的目标肽序列。诱饵肽是样本中不存在的肽段,用于评估数据的假阳性率。PQP的信息都保存在我们构建的蛋白质组学谱图库中。
第二步,基于PQP从连续采集的DIA-MS2谱图生成提取目标肽段和诱饵肽段的离子色谱图(XIC)。
第三步,碎片离子色谱图根据其肽段信息进行组和,并选择在保留时间维中具有定义峰边界的“峰组”。
第四步,对于目标肽段和诱饵肽段峰组进行打分,通过对目标肽和诱饵肽的分数分布进行统计建模来估计一组检测到的肽的错误发现率(FDR)。 一般我们控制1% FDR。
第五步,对于在大规模DIA-MS分析中,错误率控制不仅应在肽水平上进行,而且应扩展到蛋白质水平上,我们一般也会控制蛋白的错误发现率为1%。
第六步,多次运行比对校正或通过每次运行之间的色谱时间一致性匹配来增强对峰检测的置信度。
差异蛋白发现和功能注释:
针对DIA数据的后处理构建了完整的分析流程,具体包括:
(1) 第一步蛋白质定量信息的导出,将DIA原始数据导入DIA分析软件,软件自动进行DIA数据的峰匹配,同时严格控制肽段和蛋白FDR<1%,获得最终蛋白质的鉴定和定量信息;
(2) 第二步,数据质控,包括观测整体数据的动态范围等一系列的质控参数确保数据质量可靠,能用于后续的深入分析;
(3) 第三步,差异蛋白的筛选,蛋白定量结果经过t-检验或者ANOVA检验,筛选adj p-value<0.01的蛋白为差异蛋白,以火山图、PCA、OPLS-DA等统计学方法与图形化方式展示结果;
(4) 第四步,差异蛋白的功能注释及相互作用分析,差异蛋白经过GO、KEGG的注释分析获知其生物学功能,为后续的进一步功能验证提供思路。
部分统计分析结论示例如下:
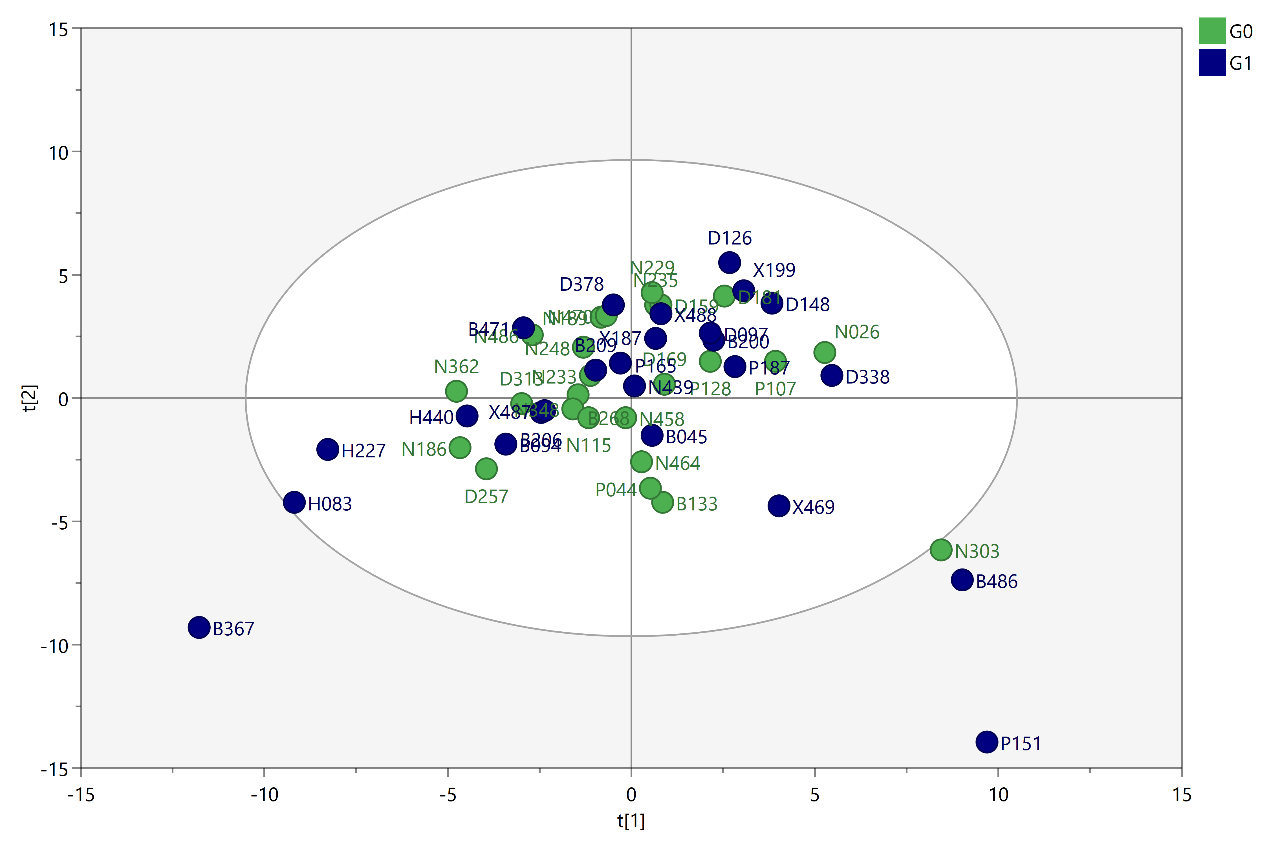
Unsupervised PCA score plots of metabolic phenotypes between G0 and G1 groups.
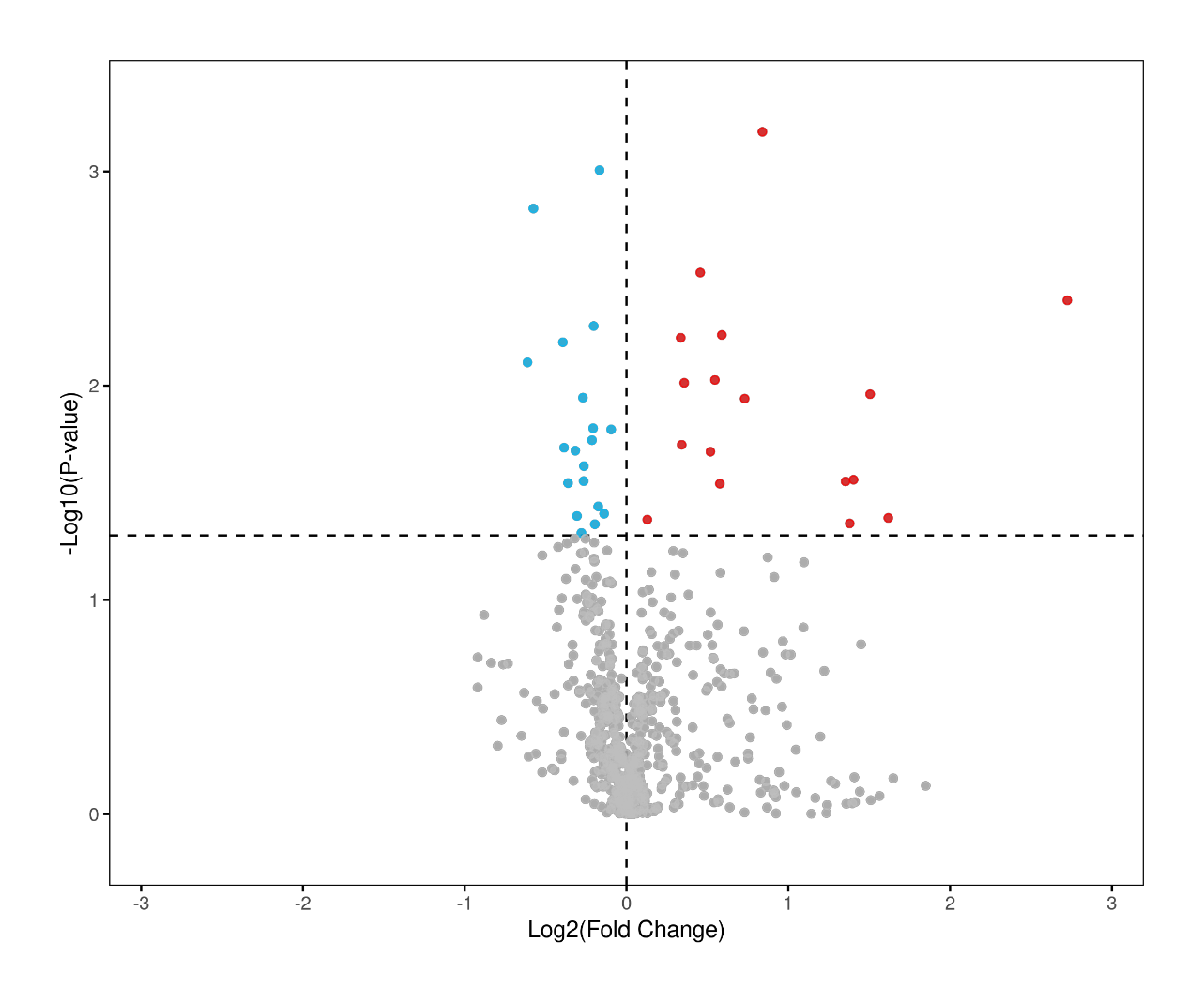
Volcano plot of quantitative DIA proteome data visualizing G0/G1.
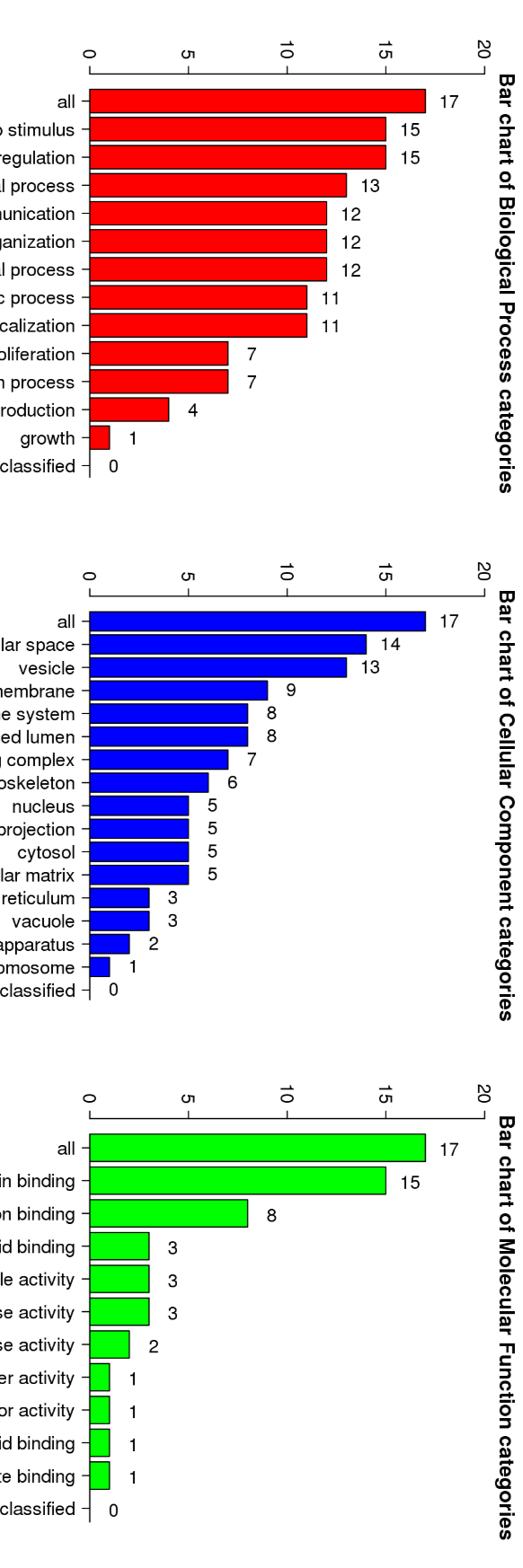
GO Slim summary for up-regulated proteins in G1 compared to G0
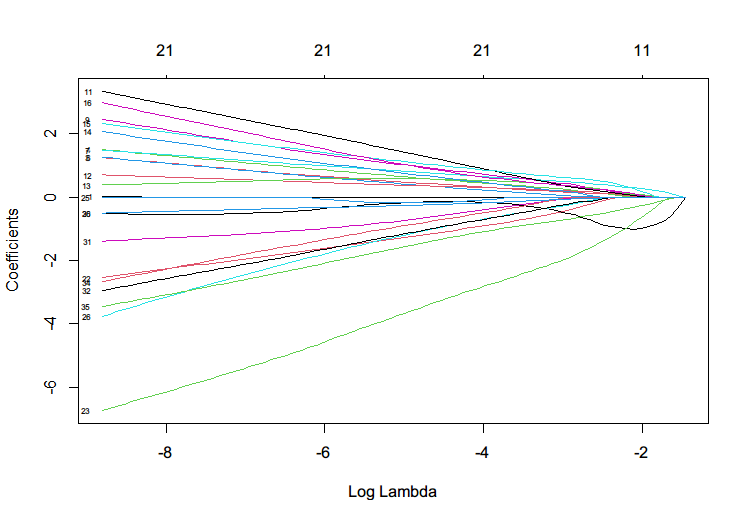
LASSO regression result between differential protein intensities with and classification information
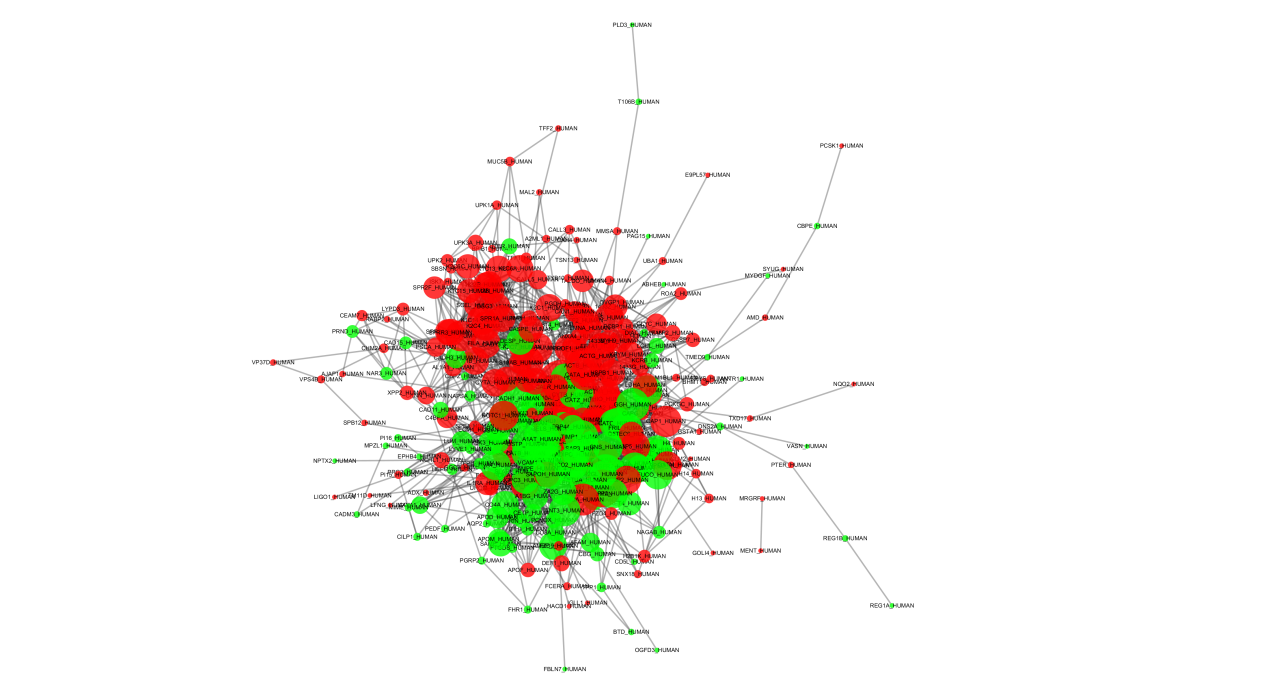
PPI 相互作用网络图
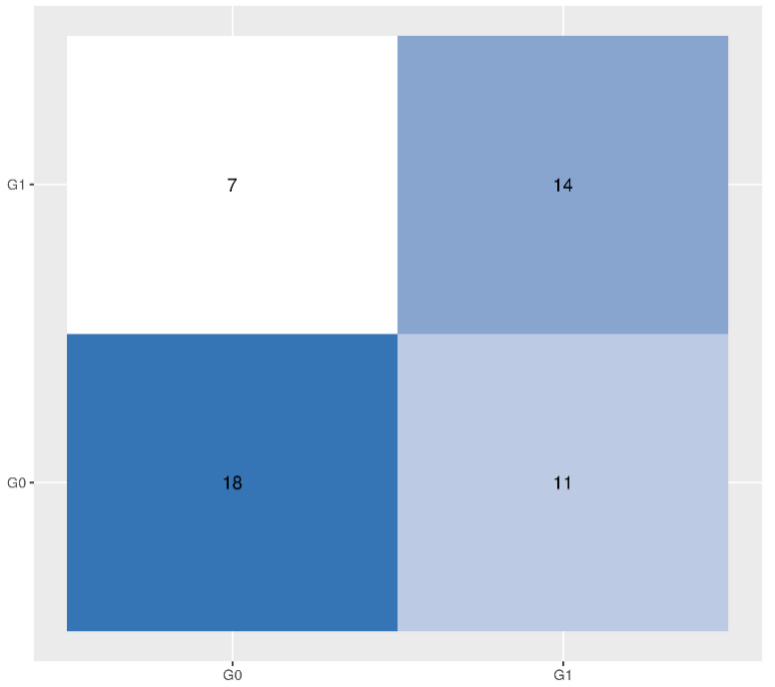
Confusion matrix of RF model (number of trees grown=150)




