多组学整合方案研究服务 - EWAS&GWAS联合研究
SNP&DNA甲基化联合研究的理论意义
SNP作为基因遗传的核心数据,具有数量丰富、分布广泛、遗传稳定等特质属性,成为复杂性状群体遗传研究的重要领域,在多组学研究体系中发挥遗传基线数据的作用,对于其他所有组学变化均存在潜在的“起因”调控,可以与不同组学的调控原件开展QTL(如eQTL、meQTL、pQTL等)关联分析、不同组学的WAS联合(如TWAS、PWAS、MWAS等)分析及MR随机化因果分析等等,属于Multi-omics研究体系中最基础的核心数据。通过上述关于长寿队列样本的GWAS研究成果调研,过往丰富的科研结论也充分证实了遗传对于长寿的影响,但是单一组学的限制均未开展深度的易感SNP位点分子调控功能机制研究,由此对于长寿的生物学机制认识片面化,也为本项目将GWAS作为辅助数据开展多组学联合方案设计提供了充足的理论支撑和创新空间!
DNA甲基化是表观基因组中最重要的组学数据,承载着个体遗传与环境变量的双重调控,对于RNA转录、剪切、转录因子结合等存在关键的调控作用,一直以来始终是系统生物学基础科研的热点方向,成为复杂表型标记物研究及表观调控机制研究的关键组学数据。充分的文献证明,DNA甲基化与年龄的相关性(DNAm Clock)是多组学中最为显著的,但是围绕长寿人群横断面的EWAS研究仍然不足,成为本项目将DNA甲基化作为多组学联合研究的核心组学设置原因之一。
近年来,伴随两个组学领域研究的深入,瓶颈问题一直制约着更具深度的基础研究体系和转化应用的发展,集中体现在:单一组学发现的易感位点对于复杂表型(疾病/性状)的调控效应微弱,无法有效构建预测模型;大部分易感位点位于非编码基因区域,或者位于非功能基因(Peripheral gene),无法有效验证其分子功能机制;单一组学更多呈现统计相关性,无法阐述因果调控机制。由此,多组学联合研究方兴未艾,从而逐步构建系统生物学完整的调控网络体系,从“管中窥豹”的独立解读上升到“庖丁解牛”的全景解读,完整呈现复杂性状的分子生物学机制。
SNP&DNA甲基化联合研究的科研目标
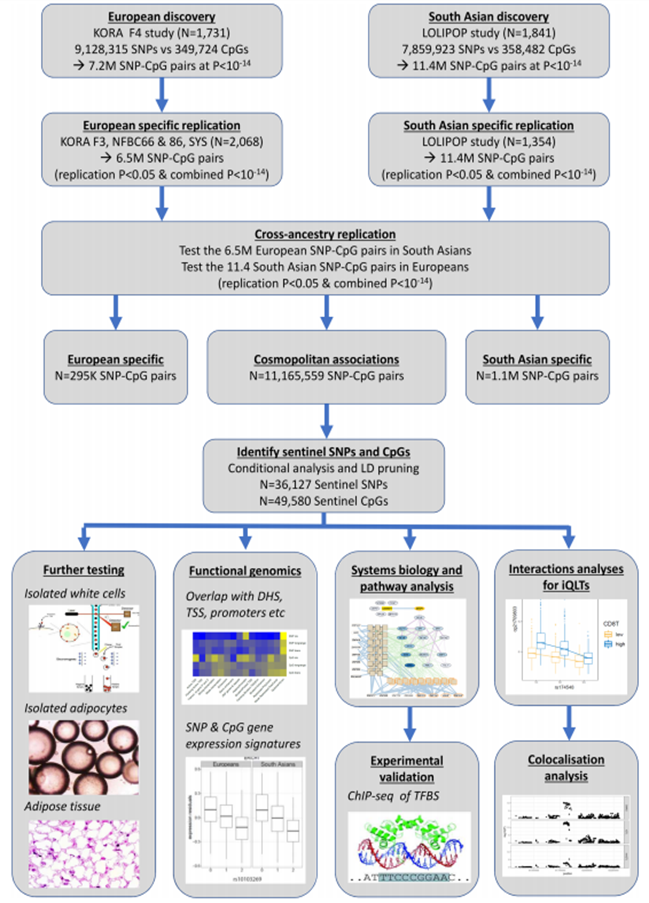
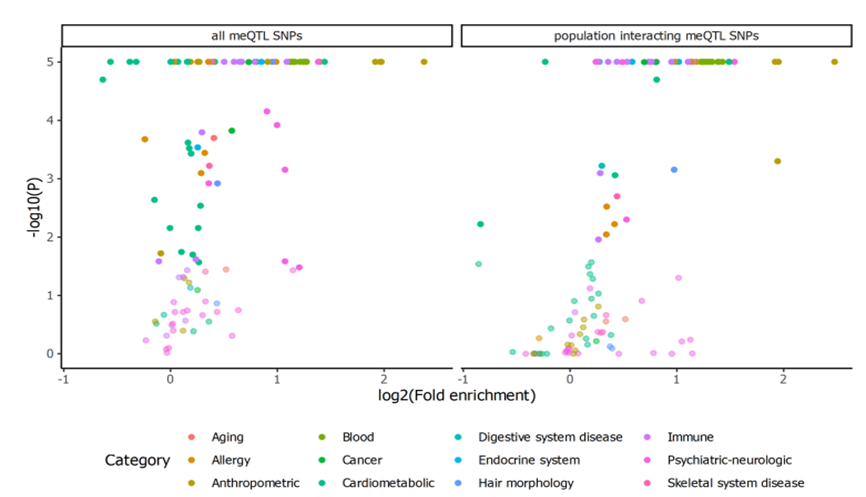
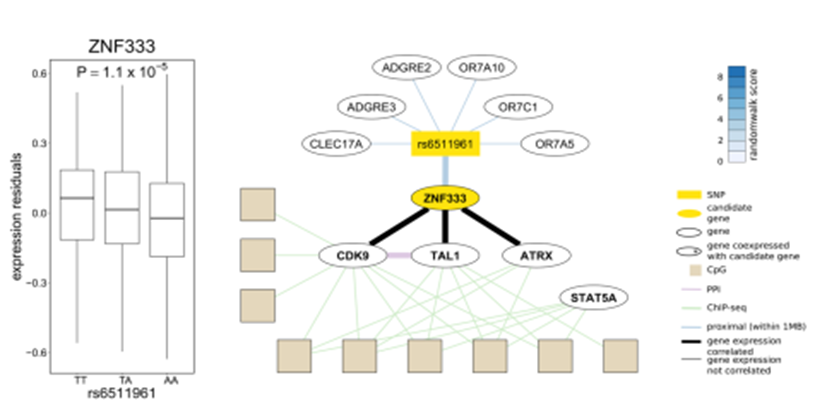
(1)构建特定人群的meQTL图谱:通过SNP与DNA甲基化的cis-及trans-关联分析,构建特定人群(如自然人群队列、疾病人群队列等)完整的meQTL图谱,探讨从遗传变异到DNA甲基化修饰的先天性调控关系,挖掘易感基因及相关富集生物通路,具有重要的科研价值,近年来发表代表性文章众多,如2022年1月份发表在Nature Genetics的文章“Genetic variation influencing DNA methylation provides insights into molecular mechanisms regulating genomic function”就是针对欧洲及南亚自然人群开展的经典meQTL,也属于目前发表的最大自然人群数量的meQTL图谱,并进行了跨种族间的对比分析。更多的关于meQTL图谱研究成果汇总请见链接:http://www.metabolomix.com/a-table-of-all-published-gwas-with-dna-methylation/。
将DNA甲基化水平(Beta值)作为样本连续性状进行关联分析,所得显著关联位点就称为mQTL(Methylation quantitative trait loci)。mQTL以分析cis-mQTL为主,trans-mQTL为辅助,其中针对候选基因的SNP与DNA甲基化联合研究则主要包括前者的关联分析,即围绕该候选基因某区域中的CpG位点甲基化水平Beta值作为因变量,联合该基因中的候选SNP位点分型结果为自变量,逐一进行各个SNP位点与甲基化位点及区域甲基化进行线性回归分析,从而筛选特定SNP与特定CpG位点显著相关性,
Cis-mQTL定义:围绕特定CpG site所属同一条染色体中(intrachromosomal)上下游各1Mb范围,共计2Mb的基因组物理区域内存在关联SNP位点,则称之为cis-meQTL。(cis-meQTLs were defined as SNPs residing within 1 Mb upstream or downstream of a CpG site. The distribution of cis-meQTLs in relation to distance from the corresponding CpGs suggested that a 2 Mb window is a reasonable window for mapping cis-meQTLs)
Trans-mQTL定义:围绕特定CpG site所属同一条染色体中(intrachromosomal)上下游1Mb范围之外的,以及不同染色体中(interchromosomal)存在关联SNP位点,则称之为trans-meQTL。(Trans-mQTLs were defined the other SNPs were analyzed for trans-acting associations for each CpG site, trans-acting regulation by factors from the genomic regions distant from the CpG sites, including from different chromosomes.)
(2)SNP与DNA甲基化中介效应分析:通过因果推断检验(Causal Inference Test),从而呈现CpG位点与SNP的因果调控关系,建立CpG作为SNP与样本表型中介调控功能机制(mediation),根据CIT的分型模型,共计包括三种类型的数据结论“methylation mediated”、“methylation consequential” 、“independent”。
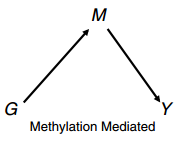
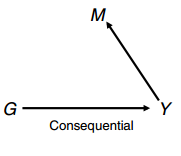
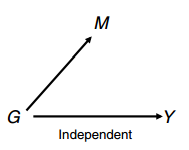
Identification of epigenetically mediated genetic risk factors for Phenotype:Genotype (G), Methylation (M), and Phenotype (Y)
(3)DNA甲基化与复杂表型MR因果效应分析:目前通过EWAS、WGBS、RRBS等技术发现的显著关联CpG site,更多呈现的是统计学意义的相关性(二分类表型、连续变量表型),却极少能够获取DNA甲基化对于表型的因果效应(CpG →Phenotype),抑或表型对于DNA甲基化的因果效应(Phenotype → CpG)。CpG不仅作为疾病重要的标记物具有重要研究意义,其因果作用的分析研究为疾病病理机制及潜在药物治疗靶点更具价值。采用Mendelian randomization (MR)分析方法,将SNP作为instrumental variables (IVs),从而分析CpG-Phenotype的因果关系。
EWAS&GWAS approach研究路径概述:
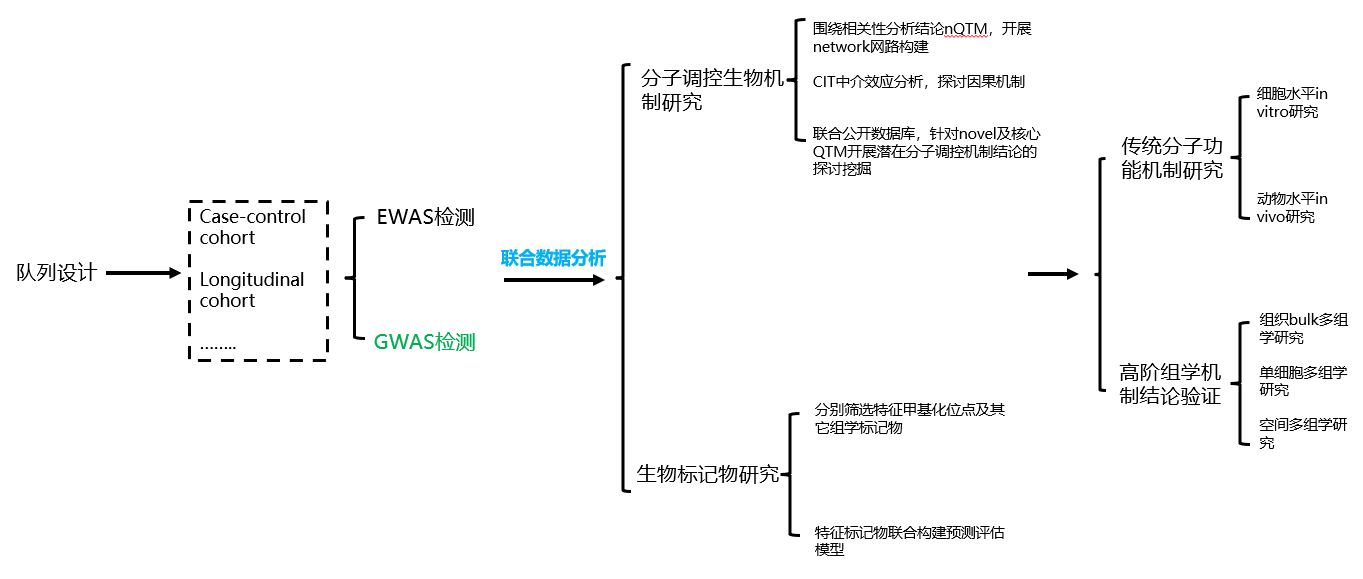
研究示例及关键统计结论示例: