代谢组生物信息学分析服务 - 代谢多组学整合数据分析服务
代谢组与GWAS整合研究
全基因组关联分析(GWAS)是研究复杂疾病遗传易感的重要技术手段,利用GWAS分析可以快速获得疾病易感的基因位点及染色体区域。由于传统GWAS筛选的位点大多数位于非功能区域,无法有效进行功能机制的深入研究,从而对于疾病的发病机理进行挖掘。因此,利用中间组学数据作为表型进行GWAS分析逐渐成为GWAS研究的热点,可以形成全面的系统生物学遗传调控机制,其中mGWAS(metabolome Genome-wide Association Study)是将代谢组数据作为表型,与基因型数据进行关联分析。由于代谢组数据对于表型鉴定更为精细,因此关联分析的精度比传统GWAS分析更高。
联合分析包括加性遗传模型、逻辑回归分析、线性回归分析等。
加性遗传模型(AdditiveGenetic Models)分析SNPs与代谢物的相关性.
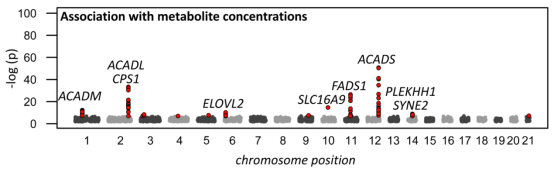
线性回归分析(linear regression)分析代谢物含量和易感SNPs间相互依赖的定量关系。
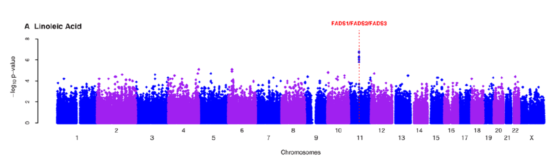
代谢组与肠道微生物基因组整合研究
在基于统计方法的关联分析中,不仅包含了基于相关性的整合分析,如皮尔森相关性分析(PearsonCorrelation)、斯皮尔曼秩相关性分析 (Spearman Rank Correlation) 等,而且还有基于数据拼接的整合分析、基于多变量的整合分析(如典型的 O2PLS 分析)和基于代谢通路(pathway)的整合分析。
代谢组学数据处理包含单变量数据分析中的T检验(Student's t test)、差异倍数分析(fold change)、方差分析(Analysis of Variance, ANOVA)、主成分分析(Principal Component Analysis,PCA)、偏最小二成判别分析(Partial Least Squares Discriminant Analysis, PLS-DA)和正交偏最小二乘判别分析(Orthogonal Partial Least Squares Discriminant Analysis, OPLS-DA)等。
肠道菌群组数据分析包含物种分类注释、T检验(T-Test)、MetaStat 分析(MetaStat analysis)、LDA Effect Size分析(LDA Effect Size analysis)、α-多样性分析(α-diversity)、β-多样性(β-diversity)、主成分分析(Principal Component Analysis,PCA)、主坐标分析(principal co-ordinates analysis),冗余分析(redundancy analysis, RDA),典范对应分析(canonical correspondence analysis, CCA),代谢通路和数据库比对分析(KEGG, CAZyme, Go)。
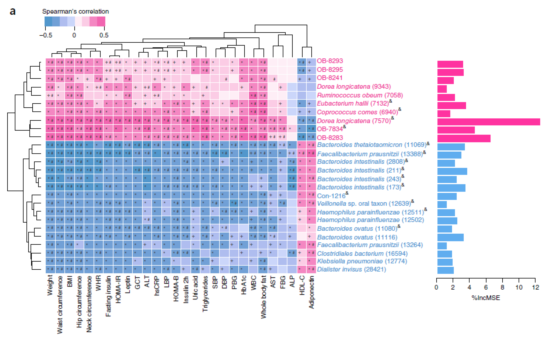
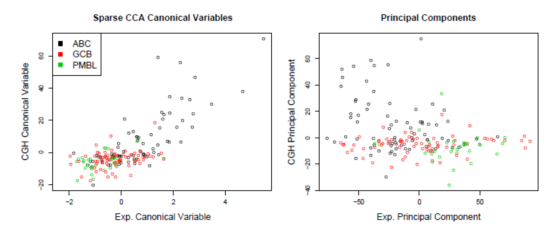
代谢组和肠道菌群的联合分析包括相关性分析(如Pearson Correlation、Spearman Rank Correlation),稀疏典型相关(Sparse Canonical Correlation)等。
皮尔森相关性分析
稀疏典型相关
代谢物来源及其相关性分析
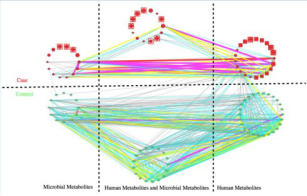
通过对代谢物进行来源性分析,主要分成三类:肠道菌群来源性代谢物,人与肠道菌群共同来源性代谢物、人体自身代谢物。对代谢物进行斯皮尔曼等级相关性分析,选取具有显著性相关作用的代谢物进行相关性展示。其中,红色原点是在疾病组中富集的差异显著性代谢物,绿色方块是在疾病组中降低的差异显著性代谢物,标记了红色外框的代谢物是研究中发现的潜在生物标记物。此外,根据斯皮尔曼等级相关性系数大小进行不同代谢物-代谢物间的相关性连接,在该图中,红色线条表示 rho≥0.9,粉色线条表示 0.9>rho≥0.8,黄色线条表示 0.8>rho≥0.7,蓝色线条表示 0.7>rho≥0.6,海蓝色线条表示 0.6>rho≥0.5,灰色线条表示 rho≤-0.5。(备注:对于菌群-菌群,菌群-代谢物都可以使用多种类似的相关性网络图展示相关性结果)
宏基因与代谢物互作网络构建
将差异基因与差异代谢物通过metscape,metmapR 等构建调控网络,该网络基于数据库、实验、文献等已有知识构建,可能能够帮助挖掘出未知的功能代谢通路,阐述新的调控机理。




