GWAS数据分析服务
发布时间:2019-04-12 分享到:
一、GWAS数据分析流程
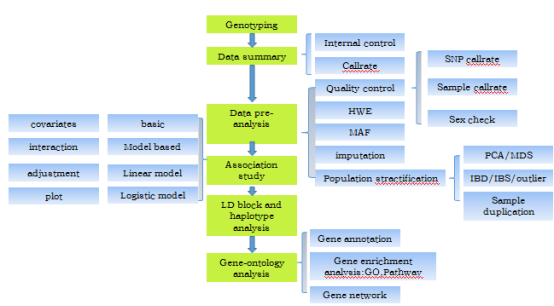
二、数据分析内容
1、分型数据质量评估:
(1)根据GWAS芯片的原始数据,通过Genomestudio V2011 genotype module V1.9.4中Manifest file及Cluster file进行分型数据质量评估,从而保证关联分析的数据质量。
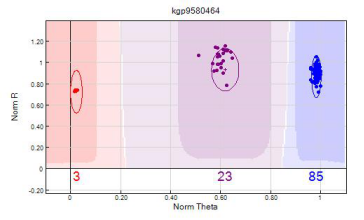
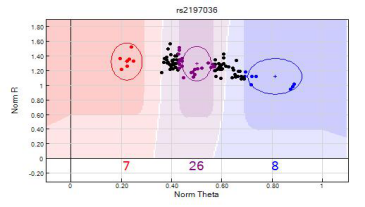
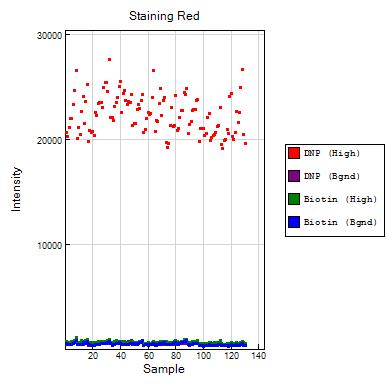
(2)根据GWAS芯片中的质控参数——Staining、Extension、Hybridization、Target Removal、Stringency、Non-specific binding、Non polymorphic等进行芯片的检测质量核实。
2、原始数据预处理:
(1)样品质控筛选:基于SNP分型数据,根据样品的callRate、分型一致率等对样品进行质量控制。剔除分型失败的样品、重复样品和可能具有亲缘关系的样品,观察样品是否污染,检验样品性别等;如将单个样本位点检测率<95%删除、将推断性别与实际性别不符的样本删除等。
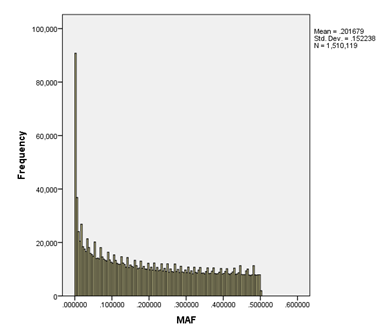
(2)位点质控筛选:对callRate、MAF、HWE检验结果等指标对SNP位点进行预处理,如将HWE<10-5不符合孟德尔遗传的位点删除、将MAF<0.01的频率过低的位点删除等。
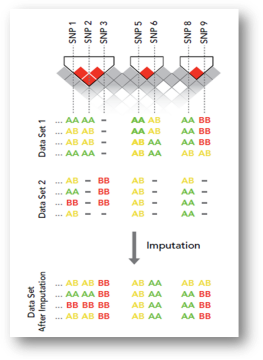
(3)Imputation分析:基于1000Genomes及Hapmap PhaseII+III中的CHB数据,采用ImptuteV2、FastPHASE、MACH等软件,针对未进行基因分型的位点之等位基因进行预测,从而有效填补GWAS的数据,增加SNP的基因覆盖度,从而使得在已经发现的关联位点周围寻找疾病位点成为可能,同时也能提高对采用了不合适的标签SNP进行标记的SNP检验效能,已经成为目前GWAS数据分析中重要的组成部分,尤其是针对特定人群采用较低覆盖度GWAS芯片进行检测,如针对中国人群采用GSA芯片的检测等。
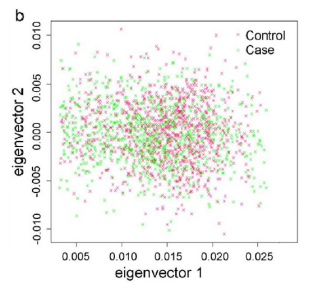
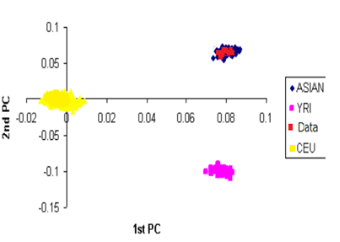
(4)人群分层分析:基于不同人群的遗传结构存在差异,为了排除人群分层对于关联分析结果影响,进行人群分层分析,进行主成分分析(principal compent analysis,PCA)或混合模型分析(mixed models)等。
3、关联分析
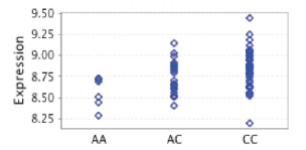
(1)Logistic/liner regression分析:通过plink软件,根据本研究中的样本的表型分组,或者样本的数量性状进行SNP的关联分析,包括imputaion的SNP位点在内,结合样本的基线数据如性别、年龄等协变量进行校正分析,进行Bonferroni、BH_FDR等多重检验,从而最大限度排除假阳性位点。
并在不同遗传模型(Allelic、Genotypic、Domiant、Recessive)进行关联分析,从而优化关联分析的P值。此外,结合样本的相关指标,进行亚组区分,并对其进行关联分析,从而分析亚分组间的异质性,及显著关联位点的区域功能关系等。
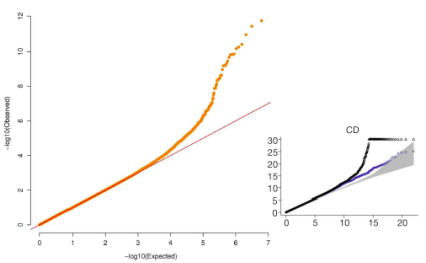
(2)QQ plot分析:基于上述关联分析的P值进行通过SPSS或Plink软件进行Quantile-Quantile Plot分析。
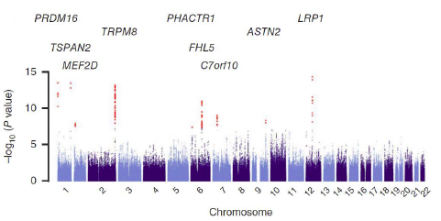
(3)Manhattan plot /Region association analysis:基于关联分析的P值,采用Python或者haploview软件进行分析。
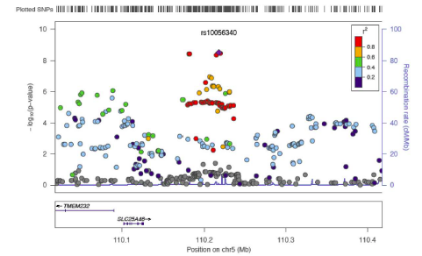
4、SNP单倍型分析
为了研究位置相邻的SNP协同作用对于样本表型或数量性状的影响,采用SHEsis/haploview软件进行单体型分析,并基于单体型进行关联分析。


5、Gene ontology分析
(1)Gene annotation(注释):根据易感SNP位于的基因及附近基因进行注释。
|
Name |
Chr |
Coordinate |
GeneSymbol |
GeneLocation |
|
VG06S52442 |
6 |
32292393 |
C6orf10 |
INTRON |
|
VG07S50448 |
7 |
117250656 |
CFTR |
CODING |
|
rs9666486 |
11 |
82449606 |
FAM181B | PRCP |
INTERGENIC |
|
kgp9732843 |
11 |
20380417 |
DBX1 | HTATIP2 |
INTERGENIC |
|
rs3740915 |
11 |
126174038 |
DCPS |
CODING |
|
rs12272467 |
11 |
5617987 |
TRIM6 / |
COMPLEX |
|
rs7478974 |
11 |
45343932 |
SYT13 | FLJ41423 |
INTERGENIC |
|
rs2276302 |
11 |
113850140 |
HTR3A |
INTRON |
|
rs1397048 |
11 |
56466099 |
LOC642975 | |
INTERGENIC |
|
rs7120582 |
11 |
11552072 |
GALNTL4 |
INTRON |
|
rs11215954 |
11 |
116348066 |
LOC100288599 | BUD13 |
INTERGENIC |
(2)Gene enrichment analysis:针对关联位点的易感基因及基因区域进行Go/pathway分析,从而推断分子调控机制的功能。
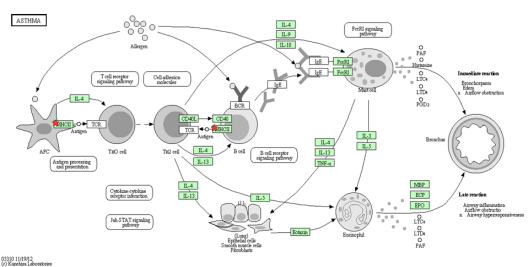
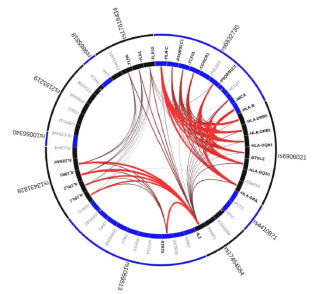
(3)Gene Network analysis:基于SNP的关联分析数据,进行基因网络调控分析,从而分析不同基因的联合效应关系。
备注:
上述分析采用的软件包括:Plink、GCTA、R packages、SNPstats、SHEsis、ImputeV2.........

相关推荐
- GWAS与EWAS联合数据分析服务2019-04-12
- GWAS数据分析服务2019-04-12




