ceRNA数据分析服务
发布时间:2019-04-10 分享到:
利用碱基序列预测microRNA-target关系对
序列数据收集
miRNA和mRNA的作用位点MRE位于mRNA的3`UTR区, 首先从UCSC数据库中得到mRNA的3`UTR序列。而miRNA也分为前体miRNA和成熟miRNA,前体miRNA需要发展为成熟miRNA才具有调节功能。成熟的miRNA序列从miRbase获得。lncRNA序列使用lncRNA芯片的原始序列。
miRanda软件
miRanda软件用来预测基因组碱基序列中的microRNA识别位点,输入文件是microRNA和需要预测的DNA或RNA的fasta文件(mRNA序列为3`UTR序列,lncRNA使用全序列)。结果包括microRNA反应元件(MREs)在microRNA上的位置、在DNA序列上的位置、MRE的自由能等信息。
结果统计
针对lncRNA或mRNA预测到的MRE数量,按照从少到多排序后做散点图。
利用表达值预测microRNA-target关系对
miRNA-target共表达
根据miRNA和target(lncRNA或mRNA)的表达值,计算出miRNA-target关系对的皮尔森相关系数。从中筛选出表达上具有负相关的miRNA-target调控关系。表达值相关系数的P值不高于0.05,且皮尔森相关系数绝对值不低于0.7,则认为具有相关性。
针对lncRNA或mRNA有相关性的miRNA数量,按照从少到多排序后做散点图。
综合碱基序列和表达值预测结果
对第一步的软件预测结果和第二步的实验验证结果求交集。实验验证提高预测的可靠性。这样,经过实验数据筛选过的miRNA-target调控关系,用于后续ceRNA的预测。
ceRNA_score原理预测ceRNA
如果lncRNA对miRNA有调控关系,而该miRNA对mRNA也有调控关系,那么该lncRAN就有可能作为该mRNA的ceRNA产生调控作用。 lncRNA作为预测的ceRNA,我们会从两个方面进行约束。lncRNA-mRNA必须有共同的miRNA,且相对高的MRE密度。
其中的ceRNA_score和pvalue的计算方法如下:
![]()
其中分子和分母表示的意义请见上表。p值计算公式如下:
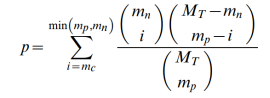
其中MT表示所有的miRNA的数量;mp表示与该mRNA有调控作用的miRNA数量;mn表示与该lncRNA有调控作用的miRNA数量;mc表示共同miRNA的数量。
利用表达值预测ceRNA
计算过程请参考第三部分。该过程是计算lncRNA和mRNA共表达。
综合ceRNA_score和表达值预测结果
通过表达值相关性计算出具有相关性的lncRNA和mRNA关系对。利用这些信息进一步过滤ceRNA_score方法预测的ceRNA。
lncRNA功能注释(KEGG、GO)
对于每一个差异表达的lncRNAs,计算得到有ceRNA关系的编码基因,用超几何分布检验方法计算每个GO或KEGG条目中差异基因富集的显著性。计算的结果会返回一个富集显著性的p值,小的p值表示差异gene在该GO或KEGG条目中出现了富集。
lncRNAs的功能预测统计特征
在上述“lncRNA名称--功能预测Term”之间的对应关系中,分别选取预测可信度(按P-value排序)最高的Top 200和Top 500个预测关系,对其中各个功能预测Term进行频次计数,统计功能注释较多的GO或KEGG term,以反映该实验中得到的差异lncRNAs功能分布的整体情况,结果见文件夹group_stat200和group_stat500中的文件。
ceRNA调控网络图
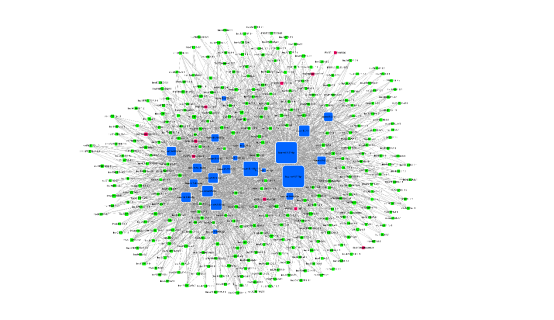
将预测的ceRNA结果关系对,按照ceRNA_score和共享miRNA进行从大到小进行排序。过滤掉共享miRNA数量过少(小于7),p值大于0.05的记录。取最可靠的前20和50条分别进行网络图的绘制。
相关推荐
- 转录组测序数据分析服务2019-04-09
- miRNA表达谱芯片数据分析服务2019-04-09
- ceRNA数据分析服务2019-04-10




